Einen globalen Wettlauf ist im Gange, um entdecken ein Impfstoff, Medikament oder die Kombination von Behandlungen, die zur Unterbrechung der SARS-CoV-2-virus, die bewirkt, dass die COVID-19-Krankheit und zu verhindern, dass die weit verbreitete Todesfälle.
Während die Forscher waren in der Lage, schnell zu identifizieren eine Handvoll bekannt, die Food and Drug Administration zugelassene Medikamente, die möglicherweise vielversprechend, weitere große Anstrengungen sind im Gange, um Bildschirm alle möglichen kleinen Molekülen, die möglicherweise die Interaktion mit den virus—und den Proteinen, die Steuern, Ihr Verhalten zu stören Ihre Tätigkeit.
Das problem ist, es gibt mehr als eine Milliarden dieser Moleküle. Ein Forscher würde in der Praxis testen möchten, die jeweils gegen die zwei Dutzend oder so Proteine in SARS-CoV-2 zu sehen, Ihre Wirkung. Ein solches Projekt könnte nutzen jedem wet-lab in die Welt und immer noch nicht abgeschlossen werden, seit Jahrhunderten.
Computer-Modellierung ist ein gemeinsames Vorgehen von akademischen Forschern und der pharmazeutischen Unternehmen als eine vorläufige, Filterung Schritt in der Medikamentenentwicklung. Jedoch, in diesem Fall, sogar jeden supercomputer auf der Erde nicht testen konnten diejenigen, die Milliarden Moleküle in einer angemessenen Höhe der Zeit.
„Ist es jemals möglich sein, allen zu werfen Rechenleistung zur Verfügung, um das problem und Holen Sie sich nützliche Erkenntnisse?“, fragt Arvind Ramanathan, ein Bioinformatiker in der Daten Wissenschaft und des Lernens-Division am U. S. Department of Energy ‚ s (DOE) Argonne National Laboratory und leitender Wissenschaftler an der University of Chicago Consortium for Advanced Science and Engineering (CASE).
Neben der Arbeit schneller, computational Wissenschaftler haben intelligenter zu arbeiten.
Eine große gemeinsame Anstrengung führte durch Forscher am Argonne vereint künstliche Intelligenz mit der Physik-basierte Droge docking und Molekulardynamik-Simulationen zu schnell zu schärfen in auf die vielversprechendsten Moleküle zu testen im Labor.
Dabei stellt sich die Herausforderung, die in ein data oder machine-learning-orientiert, problem -, Ramanathan sagt. „Wir versuchen zu bauen, die Infrastruktur zu integrieren, KI und des maschinellen Lernens Werkzeuge mit Physik-basierten tools. Wir überbrücken diese beiden Ansätze, um eine bessere bang für die buck.“
Das Projekt ist mit einigen der leistungsstärksten Supercomputer auf dem Planeten—die Frontera und Longhorn Supercomputer in dem Texas Advanced Computing Center; Gipfel am Oak Ridge National Laboratory; Theta an die Argonne Leadership Computing Facility (ALCF); und Kometen im San Diego Supercomputing Center zu laufen Millionen von Simulationen, train the machine-learning-system identifizieren Sie die Faktoren, die möglicherweise ein bestimmtes Molekül ein guter Kandidat, und führen Sie dann die weiteren Forschungen auf die vielversprechendsten Ergebnisse.
„TACC war entscheidend für unsere Arbeit, vor allem die Frontera-Maschine“, Ramanathan sagte. „Wir haben schon auf Sie für eine Weile, mit Frontera CPUs, um die maximale Kapazität zu schnell Bildschirm: unter virtuelle Moleküle und setzen Sie neben ein protein, um zu sehen, wenn es bindet, und dann Rückschlüsse, ob andere Moleküle wird auch das gleiche tun.“
Dies ist keine kleine Aufgabe. In der ersten Woche, das team testete sechs Millionen Moleküle. Sie sind derzeit die Simulation von 300.000 Liganden pro Stunde auf Frontera.
„Mit der Fähigkeit zu tun, eine große Menge von Berechnungen ist sehr gut, da gibt es uns trifft, dass wir erkennen können, für die weitere Analyse.“
Homing In Auf Einem Ziel
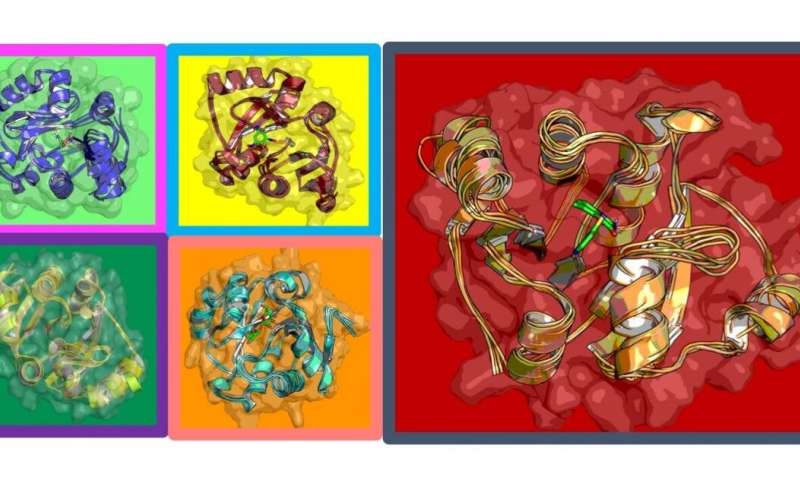
Das team begann mit der Erkundung einer der kleineren der 24-Proteinen, die COVID-19 produziert, ADRP (Adenosin-Diphosphat-ribose-1″ – phosphatase). Wissenschaftler nicht vollständig verstehen, welche Funktion das protein führt, aber es ist verwickelt in die virale Replikation.
Ihre deep-learning-plus-Physik-basierte Methode ermöglicht es Ihnen, zu reduzieren auf 1 Mrd möglich, Moleküle bis 250 Millionen Euro; 250 Millionen auf 6 Millionen; und 6 Millionen auf ein paar tausend. Von diesen wählten Sie die 30 oder so mit dem höchsten „score“ in Bezug auf Ihre Fähigkeit, binden sich fest an das protein, und stören die Struktur und Dynamik der protein—das ultimative Ziel.
Sie teilte kürzlich Ihre Ergebnisse mit experimentellen Mitarbeitern an der Universität von Chicago und der Frederick-Nationalen Laboratorium für Krebsforschung, um test im Labor und wird in Kürze veröffentlichen Ihre Daten in einer open access-Bericht, so dass Tausende von teams analysieren die Ergebnisse und Erkenntnisse gewinnen. Ergebnisse der Labor-Experimente werden weiter informieren die deep-learning-Modelle, wodurch die Feinabstimmung der Prognosen für die Zukunft-protein-Wechselwirkungen.
Das team hat sich seitdem bewegt Sie sich auf dem COVID-19 main protease, die spielt eine wesentliche Rolle in der übersetzung der viralen RNA, und bald beginnen die Arbeit an größeren Proteinen, die schwieriger zu berechnen, aber beweisen können, wichtig. Zum Beispiel, das team ist in der Vorbereitung zu simulieren Rommie Amaro ‚ s all-atom-Modell des gesamten virus, das derzeit produziert wird auf Frontera.
Die Arbeit des Teams verwendet DeepDriveMD—Deep-Learning-Driven Adaptive Molekulare Simulationen für die Protein-Faltung—eine cutting-edge-toolkit gemeinsam entwickelt von Ramanathan team am Argonne, zusammen mit Shantenu Bereich Justiz und inneres team an der Rutgers University/ Brookhaven National Laboratory (BNL), die ursprünglich als Teil des Exascale-Computing-Projekt.
Ramanathan und seine Kollegen sind nicht die einzigen Forscher, der Anwendung Maschine und Tiefe lernen, um die COVID-19 drug-discovery-problem. Aber laut Arvind, Ihr Ansatz ist selten in dem Grad, in dem KI und der simulation sind eng integriert und iterativ, und nicht nur post-simulation.
„Wir Bauten das toolkit zu tun, der Tiefe online lernen, ermöglicht es zum Beispiel, wie wir entlang gehen,“ Ramanathan sagte. „Wir üben zuerst das es mit ein paar Daten, dann lassen Sie es, um zu folgern, die eingehende simulation Daten sehr schnell. Dann, auf der Grundlage des neuen snapshots identifiziert, der Ansatz automatisch entscheidet, ob die Ausbildung muss überarbeitet werden.“
Das system legt zunächst die Bindung der Stabilität von potenziellen Molekülen in einem ziemlich einfache Art und Weise, dann fügt hinzu, mehr und mehr komplexe Elemente, wie Wasser, oder führt eine feinere Analysen der Energie-Profil des Systems. „Informationen Hinzugefügt an verschiedenen Schleusen die Punkte und auf der Grundlage der Ergebnisse könnte es überarbeiten müssen, die docking-oder machine-learning-algorithmen.“
Die komplexen workflows sind sorgfältig orchestriert über mehrere Supercomputer mit RADIKAL-Cybertools, erweiterten workload-Ausführung und scheduling-tools entwickelt, die durch computational-Experten an der Rutgers/ BNL.
„Die Arbeitsabläufe sind anspruchsvolle Anforderungen,“ sagte Shantenu Ji des rates, Vorsitzender des BNL Center for Data-Driven-Discovery und die Führung der RADIKALEN. „Dank TACC ist die technische Unterstützung, die wir erzielen konnten sowohl die gewünschten Durchsätzen und Maßstab Frontera und Longhorn innerhalb von ein paar Tagen und starten die Produktion läuft.“
Die Anwendung Der Waffen Der Wissenschaft
Das team hatte einige Vorteile, dass Ihre Forschung aus dem Boden.
Das U. S. Department of Energy betreibt einige der fortschrittlichsten X-ray crystallography labs in der Welt, und arbeitet mit vielen anderen. Sie waren schnell in der Lage, extrahieren Sie die 3-D-Strukturen von vielen der COVID-19 Proteine—der erste Schritt bei Computer-Modellierung, zu erforschen, wie solche Proteine reagieren auf Drogen ähnlichen Molekülen.
Sie waren auch aktiv an einem Projekt arbeitet, mit dem National Cancer Institute verwenden die DeepDriveMD workflow zu identifizieren vielversprechende Medikamente zur Bekämpfung von Krebs. Sie schnell geschwenkt zu COVID-19 mit tools und Methoden, die bereits getestet und optimiert.
Obwohl AI wird oft als eine black box, Ramanathan sagt, dass Ihre Methoden nicht nur blind erzeugen Sie eine Liste von Zielen. DeepDriveMD leitet, was die gemeinsamen Aspekte eines proteins machen es eine bessere Wahl ist, und kommuniziert diese Erkenntnisse, um Forscher zu helfen, Sie zu verstehen, was eigentlich passiert ist in den virus mit und ohne Medikament Wechselwirkungen.
„Unser tiefes lernen Modelle können hone in auf das Chemische Gruppen, die wir denken, sind wichtig für Interaktionen“, sagte er. „Wir wissen nicht, ob es stimmt, aber wir finden docking-scores sind höher, und es glauben erfasst wichtige Konzepte. Dies ist nicht nur wichtig für das, was passiert mit diesem virus. Wir versuchen auch, zu verstehen, wie die Viren funktionieren in der Regel.“
Sobald ein Medikament-wie kleine Molekül gefunden wird, um wirksam zu sein im Labor weitere Tests (rechnerische und experimentelle) erforderlich ist, um zu gehen von einem vielversprechenden Ziel zu heilen.
„Die Entwicklung von Impfstoffen dauert so lange, weil die Moleküle optimiert werden müssen für die Funktion. Sie müssen untersucht werden, um festzustellen, dass Sie sind nicht giftig und nicht zu tun, anderen zu Schaden, und daß Sie auch produziert werden können, im Maßstab,“ Ramanathan sagte.
Alle diese weiteren Schritte, die Forscher glauben, kann beschleunigt werden durch den Einsatz eines hybrid-AI – und physics-based modeling-Ansatz.
Rick Stevens, Argonne associate laboratory director für Computing, Umwelt und Life Sciences, TACC wurde sehr unterstützt Ihre Bemühungen.
„Die schnelle Reaktion und engagement, die wir erhalten haben von TACC hat einen entscheidenden Unterschied in unserer Fähigkeit zur Identifikation von neuen therapeutischen Optionen für COVID-19,“ sagte Stevens. „Der Zugang zu TACC computing-Ressourcen und know-how haben es uns ermöglicht, stärkt die Zusammenarbeit in der Forschung Anwendung advanced computing zu einer der heute größten Herausforderungen.“
Das Projekt Komplimente epidemiologische und genetische Forschung Bemühungen unterstützt TACC, das ist, weil mehr als 30 teams um Forschungsarbeiten, die sonst nicht erreichbar im Zeitrahmen dieser Krise erfordert.
